高斯数据拟合因 x 数据的位置而异
Gaussian data fit varying depending on position of x data
我很难理解为什么我对一组数据 (ydata) 的高斯拟合在移动对应于该数据的 x-values 间隔时效果不佳 ( xdata1 到 xdata2)。高斯写为:
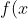&space;=&space;A&space;%5Cfrac%7B1%7D%7B%5Csigma&space;%5Csqrt%7B2%5Cpi%7D%7De%5E%7B%5Cfrac%7B-(x-%5Cmu)%5E2%7D%7B2%5Csigma%5E2%7D%7D)
其中 A 只是一个振幅因子。更改数据的某些值,很容易使其适用于这两种情况,但也可以很容易地找到它对 xdata1 效果不佳以及参数协方差不适用的情况估计的。
我在 Spyder 中使用 scipy.optimize.curve_fit,在 Windows 7.
上使用 Python 3.7.1
import numpy as np
from scipy.optimize import curve_fit
import matplotlib.pyplot as plt
xdata1 = np.linspace(-9,4,20, endpoint=True) # works fine
xdata2 = xdata1+2
ydata = np.array([8,9,15,12,14,20,24,40,54,94,160,290,400,420,300,130,40,10,8,4])
def gaussian(x, amp, mean, sigma):
return amp*np.exp(-(((x-mean)**2)/(2*sigma**2)))/(sigma*np.sqrt(2*np.pi))
popt1, pcov1 = curve_fit(gaussian, xdata1, ydata)
popt2, pcov2 = curve_fit(gaussian, xdata2, ydata)
fig, ([ax1, ax2]) = plt.subplots(nrows=1, ncols=2,figsize=(9, 4))
ax1.plot(xdata1, ydata, 'b+:', label='xdata1')
ax1.plot(xdata1, gaussian(xdata1, *popt1), 'r-', label='fit')
ax1.legend()
ax2.plot(xdata2, ydata, 'b+:', label='xdata2')
ax2.plot(xdata2, gaussian(xdata2, *popt2), 'r-', label='fit')
ax2.legend()
问题是您第二次尝试拟合高斯分布时在搜索参数 space 时卡在了局部最小值:curve_fit 是 least_squares which uses gradient descent to minimize the cost function and this is liable to get stuck in local minima 的包装器。
您应该尝试提供合理的起始参数(通过使用 curve_fit 的 p0 参数)来避免这种情况:
#... your code
y_max = np.max(y_data)
max_pos = ydata[ydata==y_max][0]
initial_guess = [y_max, max_pos, 1] # amplitude, mean, std
popt2, pcov2 = curve_fit(gaussian, xdata2, ydata, p0=initial_guess)
如您所见,它提供了一个合理的匹配:
您应该编写一个函数来提供对起始参数的合理估计。这里我只是找到了最大的 y 值,并用它来确定初始参数。我发现这对于拟合正态分布很有效,但您可以考虑其他方法。
编辑:
您还可以通过缩放振幅来解决问题:振幅太大以至于参数 space 失真,梯度下降仅遵循振幅变化最大的方向并有效地忽略 sigma。考虑参数 space 中的以下图(颜色是给定参数的拟合残差平方和,白色十字显示最优解):
确保记下 x 轴和 y 轴的不同比例。
需要在 y(振幅)中进行大量 'unit' 大小的步长才能从 x,y = (0,0) 点达到最小值,因为您只需要小于一步 'unit' 达到 x (sigma) 的最小值。该算法只是在振幅上采取步骤,因为这是最陡的梯度。当它达到使成本函数最小化的幅度时,它只是停止算法,因为它似乎已经收敛并且对 sigma 参数几乎没有变化。
解决此问题的一种方法是缩放您的 ydata 以消除参数 space 的扭曲:将您的 ydata 除以 100,您将看到您的拟合在不提供任何起始参数的情况下工作!
我很难理解为什么我对一组数据 (ydata) 的高斯拟合在移动对应于该数据的 x-values 间隔时效果不佳 ( xdata1 到 xdata2)。高斯写为:
其中 A 只是一个振幅因子。更改数据的某些值,很容易使其适用于这两种情况,但也可以很容易地找到它对 xdata1 效果不佳以及参数协方差不适用的情况估计的。
我在 Spyder 中使用 scipy.optimize.curve_fit,在 Windows 7.
import numpy as np
from scipy.optimize import curve_fit
import matplotlib.pyplot as plt
xdata1 = np.linspace(-9,4,20, endpoint=True) # works fine
xdata2 = xdata1+2
ydata = np.array([8,9,15,12,14,20,24,40,54,94,160,290,400,420,300,130,40,10,8,4])
def gaussian(x, amp, mean, sigma):
return amp*np.exp(-(((x-mean)**2)/(2*sigma**2)))/(sigma*np.sqrt(2*np.pi))
popt1, pcov1 = curve_fit(gaussian, xdata1, ydata)
popt2, pcov2 = curve_fit(gaussian, xdata2, ydata)
fig, ([ax1, ax2]) = plt.subplots(nrows=1, ncols=2,figsize=(9, 4))
ax1.plot(xdata1, ydata, 'b+:', label='xdata1')
ax1.plot(xdata1, gaussian(xdata1, *popt1), 'r-', label='fit')
ax1.legend()
ax2.plot(xdata2, ydata, 'b+:', label='xdata2')
ax2.plot(xdata2, gaussian(xdata2, *popt2), 'r-', label='fit')
ax2.legend()
问题是您第二次尝试拟合高斯分布时在搜索参数 space 时卡在了局部最小值:curve_fit 是 least_squares which uses gradient descent to minimize the cost function and this is liable to get stuck in local minima 的包装器。
您应该尝试提供合理的起始参数(通过使用 curve_fit 的 p0 参数)来避免这种情况:
#... your code
y_max = np.max(y_data)
max_pos = ydata[ydata==y_max][0]
initial_guess = [y_max, max_pos, 1] # amplitude, mean, std
popt2, pcov2 = curve_fit(gaussian, xdata2, ydata, p0=initial_guess)
如您所见,它提供了一个合理的匹配:
您应该编写一个函数来提供对起始参数的合理估计。这里我只是找到了最大的 y 值,并用它来确定初始参数。我发现这对于拟合正态分布很有效,但您可以考虑其他方法。
编辑:
您还可以通过缩放振幅来解决问题:振幅太大以至于参数 space 失真,梯度下降仅遵循振幅变化最大的方向并有效地忽略 sigma。考虑参数 space 中的以下图(颜色是给定参数的拟合残差平方和,白色十字显示最优解):
确保记下 x 轴和 y 轴的不同比例。
需要在 y(振幅)中进行大量 'unit' 大小的步长才能从 x,y = (0,0) 点达到最小值,因为您只需要小于一步 'unit' 达到 x (sigma) 的最小值。该算法只是在振幅上采取步骤,因为这是最陡的梯度。当它达到使成本函数最小化的幅度时,它只是停止算法,因为它似乎已经收敛并且对 sigma 参数几乎没有变化。
解决此问题的一种方法是缩放您的 ydata 以消除参数 space 的扭曲:将您的 ydata 除以 100,您将看到您的拟合在不提供任何起始参数的情况下工作!